유전 알고리즘(Genetic Algorithm) << 추가 작성 예정
전체 파일 확인
아래 문제의 전체 실행 결과는 여기에서 확인할 수 있다.
유전 알고리즘
유전 알고리즘은 자연계의 적자생존 원칙을 기반으로 만들어진 메타휴리스틱 알고리즘으로, 선택, 교차, 변이, 대치의 과정을 통해 점차 나은 해를 찾는 최적화 기법이다.
용어
염색체(Chromosome)
주어진 문제에서의 하나의 해. 배열 또는 문자열로 표현한다.유전자(Gene)
염색체의 개별 구성 요소. 숫자 또는 문자로 표현한다.개체군(Population)
여러 해(염색체)의 집합.인코딩(Encode)
문제 공간을 염색체의 형태로 변환하는 과정.디코딩(Decode)
인코딩된 염색체를 다시 문제 공간으로 해석하는 과정.적합도(Fitness)
각 염색체가 해의 목표에 얼마나 적합한지를 나타내는 값.
연산
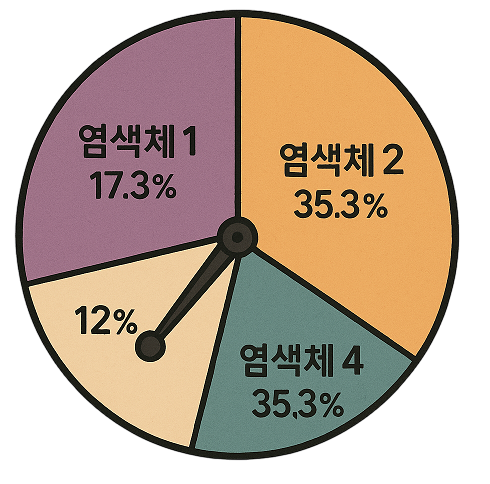
선택(Selection)
각 염색체의 적합도를 측정하여 다음 세대로 전해질 해의 후보를 선택한다.교차(Crossover)
선택된 염색체의 유전자를 결합해 새로운 유전자의 조합을 가진 후대 염색체를 만들어낸다.변이(Mutation)
후대 유전자에 무작위 변이를 도입해 지역 최적해의 빠지는 경우를 방지한다.대치(Replace)
새롭게 생성된 후대 염색체들이 기존의 염색체들을 대체함으로써 해의 질을 향상시킨다.
문제
백준 18789번을 유전 알고리즘으로 풀어볼 것이다. 푼다의 기준은 AC를 받는 조건인 8140점이상을 얻는 것으로 한다.
참고로 유전 알고리즘에서 변이 확률, 부모를 선택하는 방법, 교차 알고리즘, 대치 알고리즘, 반복 횟수 등 세부적인 알고리즘은 문제 또는 작성자에 따라 다를 수 있다.
Version 1
초기 해 생성
프로그램을 처음 실행할 때 14x8 크기의 배열을 생성한다. 개체군의 크기는 4개, 각 유전자는 0~9 사이의 랜덤한 자연수이다. 1~8140까지 등장하는 숫자의 비율에 맞춰서 초기 해를 생성해봤는데, 동일한 확률로 생성하는 것과 결과에 유의미한 차이가 없었다. 따라서 초기 해는 0~9 전부 동일한 확률로 생성되도록 하였다.
각 세대에서의 최대 적합도를 저장하는 log.txt 파일, 역대 최고의 적합도와 그 해를 저장하는 best.txt 파일, 가장 최근의 개체군을 저장하는 population.txt 파일을 사용해서 결과를 기록하고, 프로그램 시작마다 변수들을 초기화 한다.
선택
초기화된 염색체들의 적합도를 측정한다. 염색체의 적합도는 현재 문제에서의 점수로 정의한다. 적합도가 높을수록 부모 염색체가 될 확률이 높아진다.
룰렛 휠 알고리즘으로 교차 연산에서 부모로 사용할 한 쌍의 염색체를 선택한다. 각각의 염색체가 선택될 확률은 (염색체의 적합도 / 적합도의 총 합) * 100으로 구현했다.
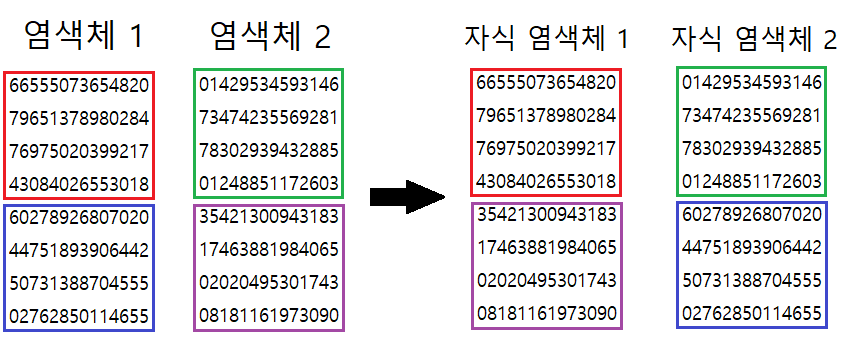
교차
이전 연산에서 선택된 부모 염색체에 교차 연산을 적용해서 자식 염색체를 만든다. 행을 기준으로 부모 염색체를 절반으로 나누고, 자식 염색체가 교차로 나눠가지는 방식으로 구현했다.
변이
일정 확률로 유전자가 변이를 일으킨다. 각 유전자가 0.05의 확률로 0~9 사이의 랜덤한 자연수로 변이하도록 구현했다. 지역 최적해에 빠지지 않기 위해서 필요한 연산이다.
대치
선택 ➞ 교차 ➞ 변이의 과정을 통해 만들어진 자식 염색체를 부모 염색체와 대치(Replace)한다.
전체 코드
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
113
114
115
116
117
118
119
120
121
122
123
124
125
126
127
128
129
130
131
132
133
134
135
136
137
138
139
140
141
142
143
144
145
146
147
148
149
150
151
152
153
154
155
156
157
158
159
160
161
162
163
164
165
166
167
168
169
170
171
172
173
174
175
176
177
178
179
180
181
182
183
184
185
186
187
188
189
190
191
192
193
194
195
196
197
198
199
200
201
202
203
204
205
206
207
208
209
210
211
212
213
214
215
216
217
218
219
220
221
222
223
224
225
226
227
228
229
230
231
232
233
234
235
236
237
238
239
240
241
242
243
244
245
246
247
248
249
250
251
252
253
254
255
256
257
258
259
260
261
262
263
264
265
266
267
268
269
270
271
272
273
274
275
276
277
278
279
280
281
282
283
284
285
286
287
288
289
290
291
292
293
294
295
296
297
298
299
300
301
302
303
304
305
306
307
308
309
310
311
312
313
314
315
316
317
318
319
320
321
322
323
324
325
326
327
328
329
330
331
#include <bits/stdc++.h>
#define X 8
#define Y 14
#define POPULATION_CNT 8 // 개체군의 크기
#define CROSSOVER_CNT 1 // 부모 염색체 쌍의 개수
#define MUTATION_RATE 0.05 // 변이 확률
#define CROSSOVER_POINT 4 // 교차 연산 시 자를 행의 기준
using namespace std;
int generation; // 현재 개체군의 세대
int best_fitness; // 모든 세대에서의 최대 적합도
int cur_best_fitness; // 현재 세대에서의 최대 적합도
int best_chromosome[X][Y]; // 모든 세대에서 최대의 적합도를 가지는 염색체
int chromosome[POPULATION_CNT][X][Y]; // 염색체
int fitness[POPULATION_CNT]; // 적합도
int parent[CROSSOVER_CNT][2]; // 부모 염색체의 인덱스
int child[CROSSOVER_CNT][2][X][Y]; // 자식 염색체
int dx[8] = {-1, -1, -1, 0, 1, 1, 1, 0};
int dy[8] = {-1, 0, 1, 1, 1, 0, -1, -1};
// 대치 연산
void Replace() {
for(int c = 0; c < CROSSOVER_CNT; c++) {
for(int i = 0; i < X; i++) {
for(int j = 0; j < Y; j++) {
// 부모 염색체와 자식 염색체를 대치
chromosome[parent[c][0]][i][j] = child[c][0][i][j];
chromosome[parent[c][1]][i][j] = child[c][1][i][j];
}
}
}
}
// 변이 연산
void Mutation() {
for(int c = 0; c < CROSSOVER_CNT; c++) {
for(int i = 0; i < X; i++) {
for(int j = 0; j < Y; j++) {
if (((double)rand() / RAND_MAX) < MUTATION_RATE) {
child[c][0][i][j] = rand() % 10;
}
if (((double)rand() / RAND_MAX) < MUTATION_RATE) {
child[c][1][i][j] = rand() % 10;
}
}
}
}
}
// 교차 연산
void Crossover() {
for(int c = 0; c < CROSSOVER_CNT; c++) {
// 부모 염색체를 { CROSSOVER_CNT, X - CROSSOVER_CNT}씩 나눠 가짐
for(int i = 0; i < CROSSOVER_POINT; i++) {
for(int j = 0; j < Y; j++) {
child[c][0][i][j] = chromosome[parent[c][0]][i][j];
child[c][1][i][j] = chromosome[parent[c][1]][i][j];
}
}
for(int i = CROSSOVER_POINT; i < X; i++) {
for(int j = 0; j < Y; j++) {
child[c][0][i][j] = chromosome[parent[c][1]][i][j];
child[c][1][i][j] = chromosome[parent[c][0]][i][j];
}
}
}
}
// log.txt 파일 업데이트
void UpdateLogFile() {
ofstream logFile("result/log.txt", ios::app);
logFile << "GENERATION [" << generation++ << "]: " << cur_best_fitness << "\n";
logFile.close();
}
// best.txt 파일 업데이트
void UpdateBestFile() {
if(best_fitness == cur_best_fitness) {
ofstream bestFile("result/best.txt");
for(int i = 0; i < X; i++) {
for(int j = 0; j < Y; j++) {
bestFile << best_chromosome[i][j];
}
bestFile << "\n";
}
bestFile << "Fitness: " << best_fitness;
bestFile.close();
}
}
// population.txt 파일 업데이트
void UpdatePopulationFile() {
ofstream populationFile("result/population.txt");
for(int p = 0; p < POPULATION_CNT; p++) {
for(int i = 0; i < X; i++) {
for(int j = 0; j < Y; j++) {
populationFile << chromosome[p][i][j];
}
populationFile << "\n";
}
populationFile << "\n";
}
populationFile.close();
}
void UpdateFiles() {
UpdatePopulationFile();
UpdateBestFile();
UpdateLogFile();
}
// crossover 대상 염색체 선택하기(룰렛 휠 알고리즘 사용)
void SelectParent() {
int totalFitness = 0;
bool selected[POPULATION_CNT] = {false, }; // 중복 선택 방지
for(int i = 0; i < POPULATION_CNT; i++) {
totalFitness += fitness[i];
}
for(int i = 0; i < CROSSOVER_CNT; i++) {
// 누적합으로 룰렛 알고리즘 구현
for(int j = 0; j < 2; j++) {
int randomValue = rand() % totalFitness;
int fitnessSum = 0;
for(int k = 0; k < POPULATION_CNT; k++) {
fitnessSum += fitness[k];
if(randomValue < fitnessSum) {
if(selected[k]) {
j--;
}
else {
selected[k] = true;
parent[i][j] = k;
}
break;
}
}
}
}
}
// p번째 염색체의 x행 y열에서 시작해서 goal[digit:end]찾기
bool FindValue(string goal, int digit, int p, int x, int y) {
if(goal.length() == digit) {
return true;
}
int value = goal[digit] - '0';
if(value == chromosome[p][x][y]) {
for(int i = 0; i < 8; i++) {
int nx = x + dx[i];
int ny = y + dy[i];
if(nx < 0 || nx >= X || ny < 0 || ny >= Y) {
continue;
}
if(FindValue(goal, digit + 1, p, nx, ny)) {
return true;
}
}
}
return false;
}
// 각 염색체의 적합도 평가
int EvaluateFitness(int p) {
int goal = 1; // 염색체에서 찾아야 하는 값
bool isExist = false; // goal이 해당 염색체에 존재하는지
while(true) {
for(int i = 0; i < X * Y; i++) {
if(FindValue(to_string(goal), 0, p, i / Y, i % Y)) {
isExist = true;
break;
}
}
if(isExist) {
isExist = false;
goal++;
}
else {
break;
}
}
return goal - 1;
}
// 선택 연산
void Selection() {
cur_best_fitness = 0;
for(int p = 0; p < POPULATION_CNT; p++) {
fitness[p] = EvaluateFitness(p);
// best_fitness 갱신
if(fitness[p] > cur_best_fitness) {
cur_best_fitness = fitness[p];
if(cur_best_fitness > best_fitness) {
best_fitness = cur_best_fitness;
for(int i = 0; i < X; i++) {
for(int j = 0; j < Y; j++) {
best_chromosome[i][j] = chromosome[p][i][j];
}
}
}
}
}
SelectParent();
UpdateFiles();
}
// best.txt에서 역대 최고 fitness값 가져오기
void InitBestFitness() {
ifstream bestFile("result/best.txt");
string line;
int x = 0;
while(getline(bestFile, line)) {
if(line.find("Fitness: ") != string::npos) {
int idx = line.find("Fitness: ") + 9;
best_fitness = stoi(line.substr(idx));
break;
}
else {
for(int y = 0; y < Y; y++) {
best_chromosome[x][y] = line[y] - '0';
}
x++;
}
}
bestFile.close();
}
// log.txt 에서 현재 세대 가져오기
void InitGeneration() {
ifstream logFile("result/log.txt");
string line;
while(getline(logFile, line)) {
if(!line.empty()) {
int startIdx = line.find('[');
int endIdx = line.find(']');
generation = stoi(line.substr(startIdx + 1, endIdx - startIdx - 1)) + 1;
}
}
logFile.close();
}
// population.txt 에서 최근 개체군을 가져와서 현재 염색체에 대입
void InitChromosome() {
ifstream populationFile("result/population.txt");
string line;
int p = 0, x = 0;
while(getline(populationFile, line)) {
if(line.empty()) {
p++;
x = 0;
}
else {
for(int y = 0; y < Y; y++) {
chromosome[p][x][y] = line[y] - '0';
}
x++;
}
}
populationFile.close();
}
void Init() {
InitChromosome();
InitGeneration();
InitBestFitness();
srand((unsigned)time(0));
}
int main() {
Init();
for(int i = 0; ; i++) {
if(i) Selection();
Crossover();
Mutation();
Replace();
}
}
결과
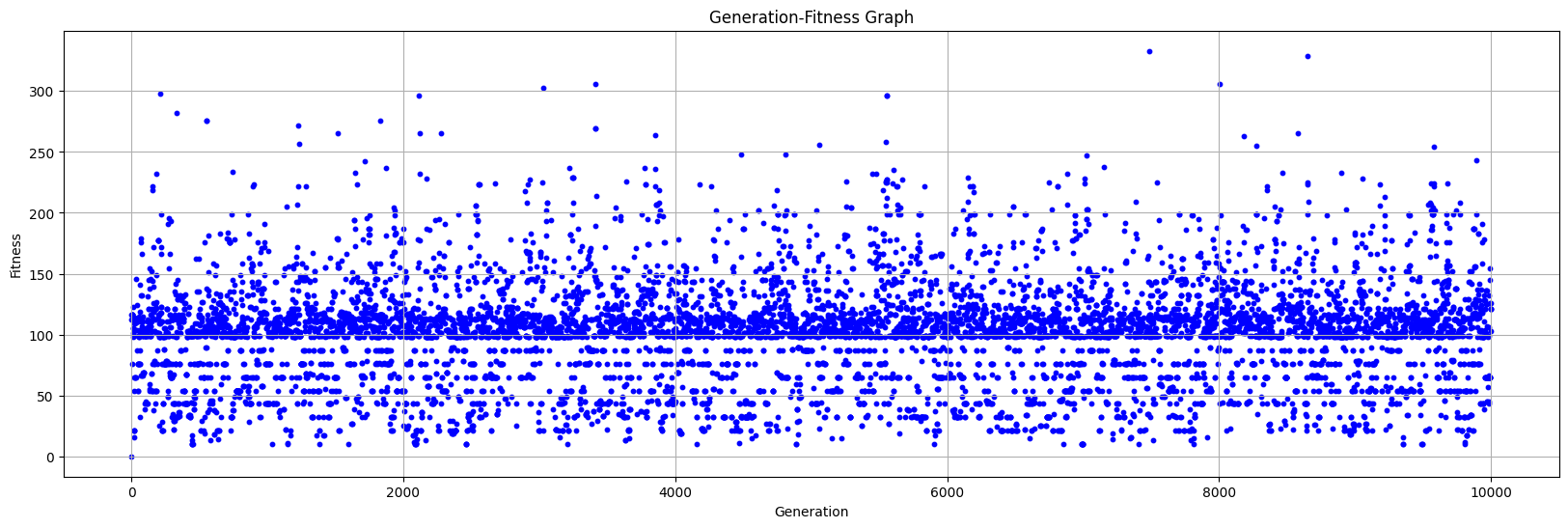
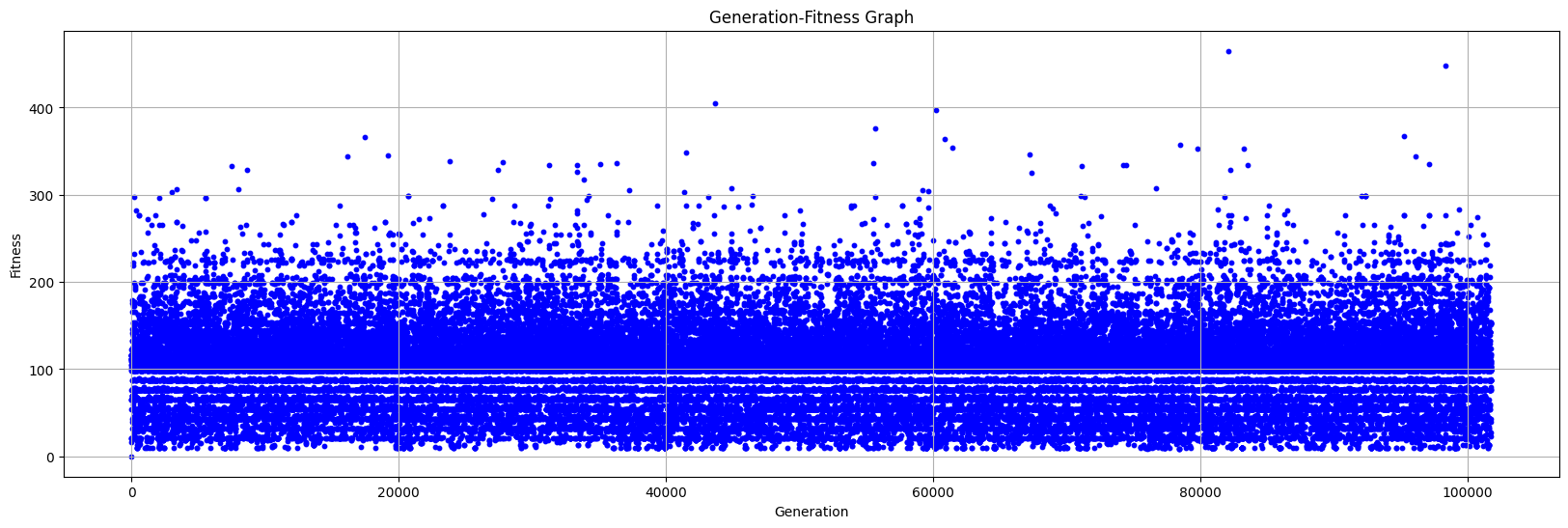
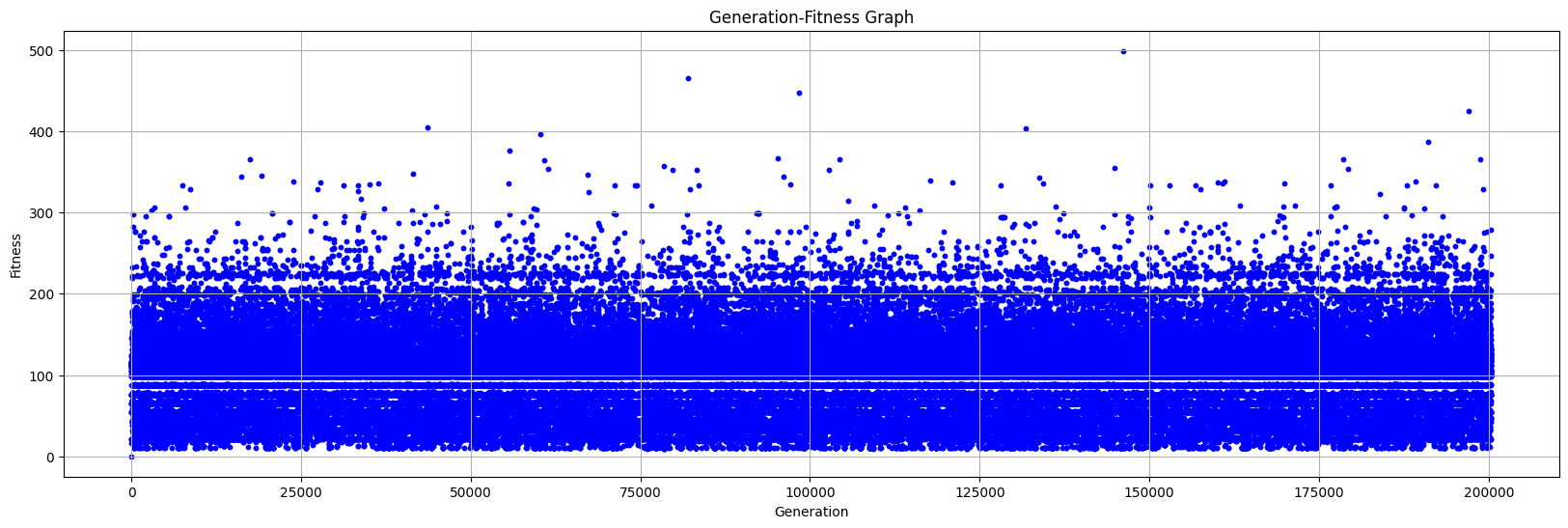
각각 1만 번, 10만 번, 20만 번 실행 결과를 표시한 그래프이다. 시행 횟수와 관계없이 일반적으로 0~200 사이에서 진동하는 것을 알 수 있다. 옛날에 자체 제작 휴리스틱 알고리즘으로 풀었을 때 2,000점은 금방 넘고 최고 4,150점까지 받아봤는데, 현재 알고리즘으로는 10억 번을 돌려도 1,000점이나 받을지 모르겠다.
개선점
교차 알고리즘
시행 횟수가 증가하면 평균 적합도도 증가해야 하는데, 지금은 항상 200 이하에서 진동하고 있다. 우수한 유전자가 후대로 전해지지 않는다는 뜻이다. 교차 알고리즘을 단순히 행을 기준으로 자르는 게 아니라, NxM 크기의 구역, 최고 적합도를 가진 염색체가 가장 덜 사용하는 구역(혹은 N개의 유전자) 등등 우수한 염색체의 유전자가 후대로 더 잘 전달되고 보존될 수 있게끔 알고리즘을 개선한다.개체군
현재 개체군에서 염색체의 개수는 8개, 교차 연산이 실행되는 염색체는 2개이다. 각각의 개수를 40개, 8개로 증가시켜서 유전자의 다양성을 늘린다.변이 확률
염색체당 유전자의 개수는 112개고, 여기에 현재 변이 확률인 0.05를 적용하면 매 변이 연산 시 평균적으로 5개 이상의 유전자가 변이한다. 숫자가 하나라도 바뀌면 적합도가 크게 달라질 수도 있는 문제의 특성상 변이 확률을 낮춰서 현재 유전자의 보존 확률을 높인다.다른 알고리즘과의 결합
SA(Simulated Annealing) 알고리즘 등 다른 알고리즘과 결합하여 Hybrid GA를 만들어 보는 방법도 있다.
Version 2
작성 예정